
Biologie Moléculaire Environnementale

Extraction des acides nucléiques
Utilisation de différents kits/systèmes manuels, semi-automatique ou automatisé pour l’extraction d’acides nucléiques (ARN et/ou ADN) à partir de différents types et volumes d’échantillons. Les méthodes peuvent être optimisées pour répondre aux spécificités des échantillons.

PCR quantitative
La qPCR (réaction de polymérisation en chaine quantitative) est une technique de biologie moléculaire qui mesure l’abondance de gènes. La mesure de l’abondance de gènes microbiens (copies de gènes par mL, par gramme, par copie de gène ARNr 16S) permet de quantifier tout marqueur d’intérêt ou la biomasse microbienne (bactérienne, virale, archéenne ou fongique).

PCR digitale
Technique de PCR de 3ème génération permettant la quantification absolue de cibles ARN ou ADN sans gamme étalon. L’échantillon et son mix de PCR sont préparés façon identique pour la PCR quantitative, puis séparés en plusieurs milliers de partitions. Chaque partition contient statistiquement 0 ou 1 séquence d’ARN ou d’ADN cible. La distribution statistique des fragments d’ADN dans les compartiments suit la loi de Poisson. Chaque partition se comporte comme un bioréacteur individuel de PCR.
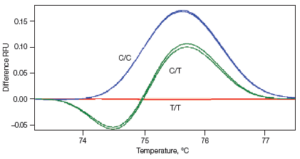
HRM
L’analyse de courbe de fusion à haute résolution (analyse HRM) est l’analyse quantitative des courbes de fusion des fragments d’ADN produits après amplification par PCR. La combinaison d’une instrumentation qPCR améliorée et de colorants de liaison à l’ADN saturants permet l’identification de petites variations dans les séquences d’acides nucléiques (polymorphismes).

Ultracentrifugeuse
L’ultracentrifugation permet de concentrer des particules virales à partir de suspensions de laboratoire ou d’échantillons environnementaux. Elle permet également de purifier des particules virales par des gradients de densité.

Ultrafiltration
L’ultrafiltration permet de concentrer les particules virales à partir de grands volumes d’eau (jusqu’à 20L) afin d’obtenir des volumes exploitables en microbiologie ou biologie moléculaire. Le rendement de concentration de cette méthode est variable selon les matrices et les virus recherchés, il peut être déterminé ou évalué.

Caméra CCD
Caméra à comptage de photons à haute sensibilité permettant l’imagerie d’émission de luminescence ou de fluorescence. La caméra est utilisable pour identifier des inductions de gènes (gènes d’éléments mobiles, gènes de virulences, …) à partir de fusions transcriptionnelles promoteur-luxCDABE ou promoteur-gfp. L’imagerie est réalisable sur des test de diffusion en gélose (type antibiogramme) ou adaptable à des objets plus complexes (réacteurs, objets biotiques ou abiotiques colonisés).

Cytomètre en flux
La cytométrie en flux permet de compter et de caractériser des cellules en haut débit à l’échelle individuelle sur des propriétés de diffusion et de fluorescence de la lumière.
Cette méthode permet la quantification de la biomasse bactérienne d’échantillons d’eau (potable, rivière), de biofilms, ou de tout autre échantillon liquide peu turbide. Après marquage avec des molécules fluorescentes (Sybr Green, Iodure de propidium, Sytox, …) ayant de forte affinité pour les acides nucléiques double brin ou simple brin, la cytométrie en flux permet de quantifier le nombre de bactéries fluorescentes totales (vivantes et mortes), leur intensité de fluorescence et selon les marqueurs utilisés différentes populations bactériennes d’un échantillon (totale et viable).
